Autor: aladelta
viernes, 21 de abril de 2006
Sección: Antropología
Información publicada por: aladelta
Mostrado 268.986 veces.
La genética y su aplicación en el estudio de las poblaciones humanas.
Se da un repaso sobre las últimas teorías sobre el origen del hombre moderno, para luego ahondar en los estudios de genética de poblaciones, haciendo hincapié en el uso del ADN mitocondrial y el cromosoma-y. Se centra finalmente y sobre todo en la población europea y muy especialmente en las poblaciones ibéricas.
Mayormente son copias de otros documentos o traducciones con algún pequeño comentario mío. Creo que afortunadamente hay en Celtiberia.net un creciente interés sobre el estudio de poblaciones mediante la genética, y espero que este articulo de una idea mejor y ayudar a interpretar mejor los documentos publicados. Ojalá se anime más la gente para que este no sea el único articulo sobre este tema.
Si alguno de vosotros desea pasarme información sobre el tema o incluso corregir algo , enviadme un correo a aladelta@yahoo.com
Saludos.
- LAS TEORÍAS SOBRE EL ORIGEN DEL HOMBRE MODERNO.
- ---EVIDENCIAS APORTADAS POR GEÓLOGOS: LA SUPEREXPLOSIÓN DEL TOBA.
- ---CONCLUSIÓN:
- ---¿SOMOS DESCENDIENTES DEL NEANDERTAL?.
- ---EL HOMO FLORESIENSIS.
- ALGUNOS CONCEPTOS QUE APARECEN COMÚNMENTE EN LAS PUBLICACIONES DE ESTUDIOS GENÉTICOS DE POBLACIONES.
- LAS HERRAMIENTAS REY DEL ESTUDIO DE LAS POBLACIONES: EL ADN MITOCONDRIAL Y EL CROMOSOMA-Y.
- ---EL ADN MITOCONDRIAL.
- ---------EL HOMBRE DE CHEDDAR.
- ---------OETZI, EL HOMBRE DE HIELO.
- ---------LAS SIETE HIJAS DE EVA.
- ---EL CROMOSOMA-Y.
- ---------VARIACIÓN EN EUROPA DE HAPLOGRUPOS DE CROMOSOMA-Y.
- EL MODELO DE DIFUSIÓN DÉMICA.
- COMPOSICIÓN GENÉTICA DE LA PENÍNSULA IBÉRICA.
- ---COMPOSICIÓN DESDE LA PERSPECTIVA DEL ADN MITOCONDRIAL.
- ---COMPOSICIÓN DESDE LA PERSPECTIVA DEL CROMOSOMA-Y.
- FENOTIPOS, LENGUAS Y GENES.
- ---FENOTIPOS.
- ---LENGUAS.
- BIBLIOGRAFÍA.
>LAS TEORÍAS SOBRE EL ORIGEN DEL HOMBRE MODERNO.
1)La Teoría Multirregional
(Wolpoff et al. 1984)
2)La Teoría del orígen africano
(Cann et al. 1987, Stringer y Andrews 1988)

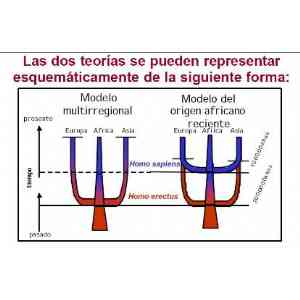
El modelo multirregional propone que el hombre moderno tiene su origen en diferentes poblaciones a partir del homo erectus, oriundo de África pero con una antiguedad de hace 2 millones de años. Estas poblaciones diferenciadas a partir de tan alejado momento daría lugar, por separado, al hombre moderno, coincidiendo con las diferentes razas actuales.
La Teoría del orígen africano es en realidad la del origen africano RECIENTE, o la Teoría de Fuera de África, la cual propone que un descendiente del homo erectus, el hombre moderno surgió de África hace unos 150.000 años y pobló todo el planeta, sustituyendo las diferentes poblaciones también descendientes del homo erectus, pero sin señales de hibridación. Una versión modificada de esta teoría es la que propone que después de la salida de África se produjo una reducción drástica de la población debido a algún tipo de evento extraordinario y que se expandió a partir de ese cuello de botella hace 50.000 años de nuevo desde África(segunda emigración). Es la llamada weak garden of eden hypothesis (WGE). Los genetistas demógrafos creen que la población humana ancestral era muy reducida --unas escasas 2.000 personas en condiciones de reproducirse, según estimaciones publicadas en diciembre de 1999.
EVIDENCIAS APORTADAS POR GEÓLOGOS: LA SUPEREXPLOSIÓN DEL TOBA.
El Toba fue un supervolcán que se encontraba en Sumatra y hace 74.000 años su caldera explotó de la forma más violenta que ha existido. Su explosión está calificada con las siglas VEI8, el punto máximo de la escala de erupciones volcánicas. Esta caldera tenía aproximadamente la superficie que tiene ahora el lago que dejó en su lugar, unos 100 km de largo y 60 km ancho. El Toba emitió gran cantidad de partículas a la atmósfera. Esto supuso un cambio climático en la Tierra durante muchos años. La radiación solar no podía llegar a la superficie porque la estratosfera estaba totalmente colapsada de particulas. La luz no penetraba para alimentar a las plantas y los animales (entre ellos nosotros) no podían alimentarse de ellas ni de otros animales porque escaseaban. Los veranos se hicieron más frescos, la nieve no se derretía y se acumulaba para el invierno siguiente, y así sucesivamente.
 |  |
|---|

El geólogo Michael Rampino estimó, con los datos acumulados por explosiones conocidas y registradas en la actualidad, que tras la superexplosión del Toba la temperatura global del planeta descendió 5ºC. Eso supuso para Europa que sus veranos sufrieran un descenso de 15 ºC. Lo cierto es que los genetistas andaban como locos intentando averiguar qué le pasó a nuestra especie para que ésta casi desapareciera hace entre 80.000 y 70.000 años. Aquí encontramos la razón. Fue un antropólogo, Stanley Ambrose, quien relacionó la explosión del Toba con la casi extinción de nuestra especie. Por supuesto que cuando ocurrió este evento, las poblaciones ya habían comenzado a colonizar el planeta y de echo se cree que los aboríenes australianos ya habían iniciado su camino hacia su actual ubicación.
CONCLUSIÓN.
La Teoría multirregional fue propuesta por paleontólogos y está sustetada por el registro fósil, mientras que a la Teoría del origen africano reciente la respalda la evidencia de los estudios genéticos cuyos resultados le da un amplio margen a su favor. A demás, las Teorías basadas en el registro fósil tiene la problemática de ser incompletas, llenas de huecos difíciles de interpretar. Por otra parte es difícil descartar completamente una Teoría multirregional más compleja con intercambio entre poblaciones, aunque para que esto pudiera haber sido así, tendría que haber habido un flujo de genes suficientes entre poblaciones geográficamente muy distanciadas a lo largo de 2 millones de años, y para ello la población debería de haber sido muy grande, permitiendo ese flujo genético entre las poblaciones, lo cual la evidencia del registro fósil no apunta en esa dirección. De todas formas, como veis ambas teorías proponen el origen del hombre moderno en África, aunque difieren en el momento en que esta emigración se llevó a cabo.
¿SOMOS DESCENDIENTES DEL NEANDERTAL?.

Los neandertales evolucionaron durante cientos de miles de años en Europa, y se apunta a que quedaron aislados del resto de homínidos, mientras que nuestros antepasados se cree que todavía no habían salido de África.

Sobre si el neandertal y nuestra especie pertenecían a la misma especie se realizó un estudio en el cual se pudo extraer una muestra de ADN de un neandertal apta para el análisis y compararlo con los del hombre moderno. El ADN no fosiliza y, aunque lo encontremos, en la mayoría de las veces estará contaminado. Aún así, se pudo extraer una muestra de ADN "sin contaminar" del interior de un hueso fosilizado de neandertal y analizarlo. El resultado de este análisis demostró que su ADN es muy parecido al nuestro, pero distinto, demostrando que somos dos especies emparentadas(ambas especies descienden del homo antecessor) pero diferenciadas por el aislamiento mútuo. Nuestro linaje y el de los neandertales se separó hace entre 550.000 y 690.000 años, mientras que nuestra diversidad actual surge entre hace 120.000 y 150.000 años.


Un híbrido potencial Neandertal/sapiens de hace 24,500 años proveniente del sitio Lagar Velho, en Portugal, fue anunciado.
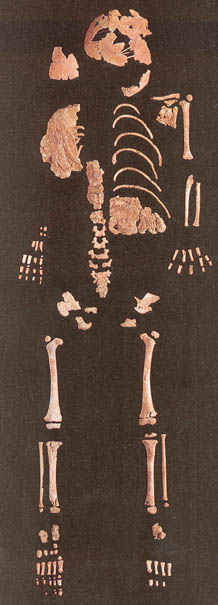
Este individuo de 4 años de edad tiene un cuerpo corto y ancho como un Neandertal, pero posee un cráneo anatómicamente moderno. Existe una serie de problemas en la interpretación de este hallazgo como un híbrido Neandertal/sapiens. Primero que todo, como un híbrido, debería tener una mezcla de caracteres en todo el cuerpo y no simplemente poseer el cuerpo de un Neandertal y el cráneo de un humano moderno. Por ejemplo, si observamos a los híbridos entre leones y tigres, ellos no poseen la cabeza de una especie y el cuerpo de la otra, sino que exhiben una mezcla morfológica de ambas especies. Segundo, y más importante, la aceptación de este espécimen como un híbrido sugeriría que los caracteres de los Neandertales fueron retenidos por entre 6,000 a 10,000 años después de que los Neandertales se extinguieron, lo cual es muy poco probable. Esto es teóricamente improbable porque los caracteres de los Neandertales habrían sido completamente diluidos por los genes de Homo sapiens en un periodo de tiempo tan largo.
La paleontología y la genética, de todas formas, parece dejar la puerta abierta a ésta hibridación entre neandertales y hombres modernos, pero para demostrar eso se deben encontrar fósiles y conseguir ADN no contaminado para compararlo con la de los hombres modernos, y de momento no se ha conseguido.
Enlace a EL ÚLTIMO NENDERTAL
Para saber más sobre el niño de Lagar Velho (Portugal) pincha aquí
EL HOMO FLORESIENSIS.

En septiembre de 2003 científicos indonesios y australianos hicieron un increíble descubrimiento en la isla de Flores, Indonesia. Encontraron enterrados en una cueva un esqueleto de lo que parecía un niño, por su poca estatura, alrededor de un metro. Sin embargo, después de examinarlo, se dieron cuenta de lo que tenían delante no era sino un homínido adulto hembra. Los restos tienen una edad de 18.000 años y tras examinarlos detenidamente se llegó a la conclusión de que era una especie nueva descendente probablemente de una variante del homo erectus que se estima llegó a la isla hace aproximadamente un millón de años evolucionando en ella hasta alcanzar estas características. Era bípedo y caminaba erguido, usó herramientas de piedra sofisticadas, y parece que hasta supo hacer fuego, el cual utilizaba para cocinar la caza de la que se alimentaba, ¡todo eso con una capacidad craneana de 380 cc, parecido al tamaño del cerebro de chimpancé!, lo cual desmonta la teoría de la necesidad de un cerebro grande para el desarrollo de la inteligencia. Se cree que se extinguió hace unos 12.000 años a raíz de una erupción volcánica que devastó la isla, o que perdió la batalla contra otros grupos homo.

Lo más intrigante de todo esto es que los habitantes de la isla cuentan leyendas de unos seres de las mismas características que llamaban Ebu Gogo, los cuales se murmuraban los unos a los otros en lo que parecía un lenguaje, repitiendo como loros lo que decía el otro, a modo de colación. ¡La última leyenda data de 100 años!. Henry Gee, el editor de la revista Nature se atreve incluso a especular que estas criaturas puedan todavía existir en algún lugar de zonas vírgenes de indonesia.

Alguien podría pensar si el homo floresiensis tiene algo que ver con los pigmeos. Rotundamente no. Los pigmeos pertenecen a nuestra misma especie, son nuestros parientes directos, nuestros hermanos de sangre; son señores bajitos y cabezones, con una capacidad craneana comparable a la del resto de humanos modernos. Mientras que el homo floresiensis es nuestro primo lejano, que aunque vivió al mismo tiempo que nuestra especie pertenece a una especie inteligente, pero diferenciada de la nuestra.

El principio de coalescencia: Asume la existencia de un origen común a todos los seres vivos. Por este mismo principio se asume un origen común a todos los seres humanos.
Marcadores clásicos: Las investigaciones se centraban en el estudio de antígenos eritrocitarios (sistema ABO, Rh, MN), proteínas séricas, enzimas eritrocitarias y sistema HLA.
RFPL (Restriction Fragment Length Polimorphism):Análisis de polimorfismos de longitud de fragmentos de restricción.
HLA (human leukocyte antigen): Antígenos de los leucocitos humanos. Determinan la compatibilidad de los transplantes.
LGM (Last Glacial Maximum): La máxima expansión de los hielos durante la última glaciación se calcula que ocurrió en el 18.000 a. C. aproximadamente. La última glaciación o glaciación de Würm ocurrió entre el año 35.000 a. C. Y el 10.000 a.C aprox.
Microsatélites o STRs (Short Tandem Repeat polymorphisms). Son secuencias de ADN repetidas en tandem de 2 a 6 pares de bases. Se emplean como marcadores genéticos para rastrear la herencia familiar o mapear enfermedades en el genoma. La tasa de mutación es grande, por lo que se utiliza para conocer eventos demográficos recientes ocurridos en una escala de tiempo más reducida.
Minisatélites o VNTR (variable number of tandem repeats). Son repeticiones también en tandem. Se diferencian de los microsatélites en que las secuencias que se repiten son mayores.
Polimorfismos de inserción alu. Las inserciones Alu son elementos polimorficos de aproximadamente 300 pares de bases, que se encuentran distribuidas en el genoma de los primates. La especie humana posee incorporada en sus celulas su propia subfamilia. La inserción polimórfica en lugares específicos del genoma de estos elementos Alu, sirven para el estudio de la historia demográfica de las poblaciones. Sin embargo el grado de polimorfismo es bajo (sólo hay dos alelos, que en realidad son presencia y ausencia de inserciones en las secuencias). Tampoco se pueden construir filogénias, por lo que se limita su aplicación para el estudio de poblaciones.
Mutaciones puntuales o SNPs (Single Nucleotid Polymorphism). Un SNP se define como la presencia de dos posibles bases en una posición particular en el ADN. Esta posición particular debida a mutaciones puntuales constituyen la principal contribución a la variación genética humana y cuya densidad en el genoma es del orden de 0,5-10 por 1000 pares de bases. El interés de los SNPs reside en su posible asociación con el desarrollo de enfermedades y en las enormes posibilidades que ofrecen para el descubrimiento de la base genética de la susceptibilidad a las enfermedades complejas o de la sensibilidad a los agentes terapéuticos (farmacogenética). Marcador bialélico. Asociado con la porción del cromosoma-y que no se recombina en la fecundación.
Alelos de diferentes genes (HLA entre otros). Un alelo es una de las formas variantes de un gen en un locus(lugar del cromosoma donde está localizado un gen específico) o de un marcador particular en un cromosoma. Diferentes alelos de un gen producen variaciones en las características hereditarias tales como el color del cabello o el tipo de sangre.
NRY: Non-Recombining region of Y chromosome. Región del cromosoma-y que no se recombina durante la fecundación.
Fenotipo: El fenotipo de los organismos comprende las características observables directamente por nuestros sentidos(morfología), que se originan como consecuencia de las interacciones entre el genotipo y el ambiente.
Alelo dominante y alelo recesivo: Son dos formas de un mismo gen que codifican para caracteres determinados. El alelo dominante es el que, estando en uno solo de los cromosomas de cada pareja de un individuo, se manifiesta en el fenotipo de éste. El recesivo es el alelo que debe estar presente en los dos cromosomas para que se manifieste en el fenotipo el carácter para el que codifica.
ADN (el): Abreviatura de ácido desoxirribonucleico, constituyente esencial de los cromosomas del núcleo celular.
Genoma: El material genético de un organismo.
Mitocondria: En el citoplasma de las células con núcleo diferenciado, orgánulo encargado de la obtención de energía mediante la respiración celular.
Cromosoma: Cada uno de los filamentos de material hereditario que forman parte del núcleo celular y que tienen como función conservar, transmitir y expresar la información genética que contienen: El ser humano tiene 23 pares de cromosomas.
Haploide(adj): Referido a un organismo o a su fase de desarrollo, que tiene una dotación simple de cromosomas. Los óvulos y los espermatozoides son células haploides, pero un embrión es ya diploide.
Filogenia: La evolución de un grupo de organismos genéticamente relacionados, para diferenciarla del desarrollo de un organismo individual.
Marcador: También conocido como marcador genético, es un segmento de ADN cuya herencia se puede rastrear. Un marcador puede ser un gen, o puede ser un segmento de ADN sin función conocida. Dado que los segmentos de ADN que se encuentran contiguos en un cromosoma tienden a heredarse juntos, los marcadores se usan a menudo como formas indirectas de rastrear el patrón hereditario de genes que no han sido aún identificados, pero cuyas ubicaciones aproximadas se conocen.
Haplotipo: tipo de secuencia que comprende todas las secuencias idénticas. Estas secuencias están compuestas por combinaciones alélicas.
Haplogrupo:grupo de haplotipos que comparten un ancestro común. Es decir, estos haplotipos forman un conjunto que se repite en una población, y que define el haplogrupo.
Hg: Haplogrupo
MtDNA: Ácido desoxiribonucléico(ADN) mitocondrial o ADNmt.
Me dejo otras muchas definiciones, pero “grosso modo” están las más importantes para tener una idea general.
Los linajes que resultan como consecuencia de mutaciones, tanto en el ADN mitocondrial como en el cromosoma-y son conocidos como haplogrupos. Cada haplogrupo lo conforman diversos haplotipos que lo definen.
Las mutaciones que producen la variación de haplotipos dan como resultado un nuevo subhaplogrupo. Todos los seres humanos compartimos haplotipos comunes, que definen el haplogrupo común, nuestro Adam del cromosoma-y, o nuestra Eva del ADN mitocondrial. A partir de éste haplogrupo común surgen otros haplogrupos diferenciados por variaciones de estos haplotipos, y así sucesivamente.
EL ADN MITOCONDRIAL.
El ADN mitocondrial no se encuentra en el núcleo de las células, sino en unos orgánulos del citoplasma (es decir, fuera del núcleo) que se llaman mitocondrias. Cuando se produce la fecundación del óvulo, el espermatozoide aporta la mitad de los cromosomas. La otra mitad los pone el óvulo. Pero sólo la madre proporciona el ADN mitocondrial, ya que el espermatozoide no contribuye con mitocondrias(la cola del espermatozoide posee mitocondrias, pero se pierde durante la fecundación del óvulo).
En principio, todas las personas deberían tener la misma cadena de letras de ADN en sus mitocondrias. En la realidad, el ADN mitocondrial ha acumulado progresivamente cambios durante milenios debido a errores de copia y a daños por radiación. Es por eso que algunos cambios sólo aparecen en regiones o continentes específicos. En un artículo publicado en marzo del 2000 en The American Journal of Human Genetics, Douglas C. Wallace y algunos colegas identifican a los Vasikela Kung, del noroeste del desierto de Kalahari, al sur de África, como el grupo racial que está más próximo a la raíz del árbol del ADN mitocondrial humano. Otro grupo racial que parece casi igualmente antiguo es el de los pigmeos Biaka del África Central.
El árbol del ADN mitocondrial está enraizado en un único individuo, la Eva mitocondrial, porque todos los otros linajes se extinguieron.
El primero en utilizar la Herramienta del ADN mitocondrial fue Wesley Brown en 1980. Pero el que realmente tuvo repercusión popular fue el equipo de Allan Wilson en 1987. Ambos, sin embargo, llegaron a las mismas conclusiones, esto es: el origen común de todos los seres humanos en un linaje no más antiguo que 180.000 años. Luego vendrían otros complementando a los anteriores, el grupo de Cann en el 87 con su Eva mitocondrial basado en el principio de coalescencia, Vigilant y colaboradores en el 91, corroborando el anterior, así hasta más tarde desentrañar los diferentes haplogrupos a partir del original.
Dejando de nombrar ya un poco a los muchos autores que han estudiado y definido los diferentes haplogrupos, empezaremos a poner nombre a estos:
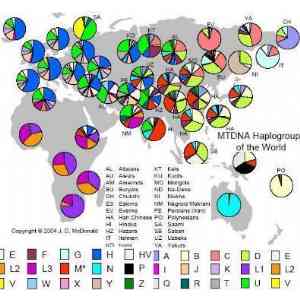
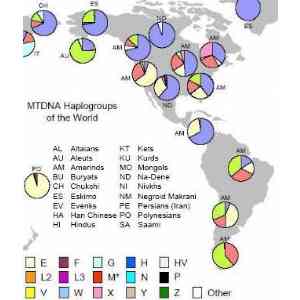
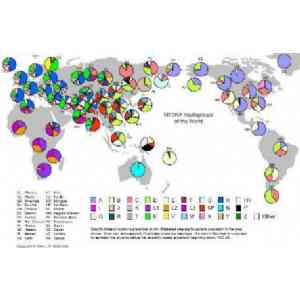
Macrohaplogrupo L

Pinchar aquí para ver la expansión de mtDNA mundial según J. D. Mcdonald, por pasos.
(nuestra Eva mitocondrial común), que ha su vez se subdivide en L0, L1, L2, L3, M y N. Los subhaplogrupos L0, L1, L2 son específicos del África subsahariana, mientras que los M y N aparecidos al noreste de África, se expandieron por Europa y Asia.
Los haplogrupos que poblarán Asia surgen tanto del macrohaplogrupo M (que son los denominados C, D, E, G, Z) y el macrohaplogrupo N (subdividido en A, B, F, Y)
Los haplogrupos que poblarán América (antes de las invasiones europeas) son el A, B, C, D y X constituyendo el 100% de linajes.
Los haplogrupos que poblarán Europa surgen del macrohaplogrupo N y son: H, I, N1b, T, U, V, W y X. Estos haplogrupos constituyen el 98% de los linajes europeos.
EL HOMBRE DE CHEDDAR.

En 1903, en una cueva cerca de Cheddar, en el sur de Inglaterra, se encontró enterrado un esqueleto no fosilizado de un hombre. Se determinó que el esqueleto tenía una antigüedad de 9.000 años. Bryan Sykes analizó una prueba de ADN mitocondrial de una muela del esqueleto. Su ADN fue comparado con el de los habitantes de Cheddar. Los resultados fueron impactantes. El hombre de Cheddar, que había vivido hacía 9.000 años, resultó tener un pariente directo vivo en Cheddar, un profesor de escuela. El test también sirvió para demostrar que los britones no eran descendientes de emigrantes neolíticos venidos de Oriente Medio, como se había creído tradicionalmente, sino que estos eran descendientes de cazadores recolectores que llegaron a las islas hace aproximadamente 10.000 años.
OETZI, EL HOMBRE DE HIELO.
En 1991 dos turistas alemanes encontraron lo que parecía los restos de un ser humano momificado y conservado por deshidratación, el frío y la nieve que lo cubría, que se tornó en hielo durante los más de 5.000 años que estuvo enterrado en él; estaba atrapado en hielo glaciar, en los Alpes tiroleses, cerca de la frontera entre Alemania y Austria. Junto al cuerpo había un hacha de cobre, una daga, un arco y 14 flechas. El estudio forense evidenció que el individuo al que se estimó de 46 años de edad en el momento de su muerte y de 1.59 m de altura, fue herido de muerte por una flecha que le entró a la altura del pecho y murió poco después desangrado. Algunos “alumnos aventajados” dijeron que se trataban de un fraude, una momia india americana falsa, plantada a propósito en el hielo glaciar. Así que tras remover tejido del cuerpo por los forenses para ser analizado mediante la prueba del ADN mitocondrial por el equipo del profesor Bryan Sykes se llegó a la conclusión de que no estaba emparentado ni con ningún nativo americano, ni con siberianos, ni subsaharianos, sino que lo estaba con los habitantes que habitaban hoy en día en los Alpes. Un grupo de investigadores de la Universidad de Queensland, Australia, determinó que el hombre de hielo nació en lo que ahora es el pueblo de Velturno(Feldthurns), en el sur del tirol Italiano (norte de Italia). Se ha encontrado incluso un pariente actual de Oetzi, que vive en Dorset, sur de Inglaterra; es una irlandesa que dio la casualidad que trabajaba en el laboratorio de Sykes. Los científicos, además, lograron precisar que el hombre de hielo pasaba los veranos en las montañas de Vinschgau, al norte de su pueblo natal.
 |  |
|---|
La conclusión a la que se llegó, gracias a la prueba genética y la del carbono 14, (isótopos hallados en los huesos y dientes) y la composición química de muestras de agua y tierra de un área de los Alpes, fue que el hombre de hielo, al que llamaron Oetzi, era un cazador neolítico que murió intentando cruzar los Alpes hace 5.300 años y que esta emparentado con los habitantes actuales de esa zona, además de con el resto de europeos, lo que además demuestra la estabilidad del ADN para su aplicación al estudio de poblaciones.
Para saber más, pincha aquí.
LAS SIETE HIJAS DE EVA.
A raíz del estudio de Oetzi, Sykes decidió, movido por la gran curiosidad de encontrar parentescos entre los seres humanos, dejar su carrera de genetista médico para concentrarse en el estudio de las poblaciones.
En el 2001 publicó “las siete hijas de Eva”. En él, además de hablarnos del hombre de hielo, nos revela que a partir de sus estudios realizados durante una década, ha descubierto que la mayoría de europeos desciende de sólo siete linajes de ADN mitocondrial, o como a él le gusta llamarlas, las siete hijas de Eva. Además les puso nombres inspirados en la mitología europea:
Ursula(osa en latín): Se corresponde con el haplogrupo U5. Es el linaje más viejo de los siete. Se remonta a 45.000 años con los primeros humanos modernos. El 11% de los europeos descienden directamente de ella. Extendido por toda Europa, es especialmente frecuente en el oeste de las Islas Británicas y Escandinavia.
Xenia(hospitalario en griego): Se corresponde con el haplogrupo X. Es el segundo linaje más antiguo de los siete. Fue fundado hace 25.000 años por la segunda oleada de hombres modernos, justo antes de la llegada de la última glaciación. El 7% de los europeos pertenecen a este linaje. Se subdivide en tres subhaplogrupos; uno extendido por el Este de Europa y los otros dos por el Centro. Curiosamente el 1% de los indígenas americanos forman parte de este grupo.
Helena(luz en griego). Se corresponde con el haplogrupo H. Es, con diferencia, el linaje más extendido en toda Europa con un 41% de europeos que pertenecen a este linaje. Este se originó hace 20.000 años en algún lugar entre los valles de Dordoña y el norte de la Península Ibérica. Como ya he dicho, está extendido por toda Europa, aunque su frecuencia más alta está entre los vascos. Su expansión se inició después de la retirada de los hielos, al finalizar la última glaciación.
Velda(soberana en escandinavo). Se corresponde con el haplogrupo V. Es el linaje más modesto de los siete con sólo un 4% entre europeos descendientes directos. Velda vivió inicialmente en el norte de la Península Ibérica hace 17.000 años. Sus descendientes habitan principalmente en el oeste y norte de Europa siendo curiosamente muy frecuentes entre los lapones, habitantes de Finlandia y norte de Noruega.
Tara(colina rocosa en gaélico). Se corresponde con el haplogrupo T. Aproximadamente el 10% de los europeos pertenece a este linaje. Se expandió desde el Norte de Italia entre las colinas toscanas y las rías del río Arno hace 17.000 años. Su principal expansión se centra en el sur y oeste europeos, alcanzando grandes concentraciones en Irlanda y oeste de Gran Bretaña.
Katrine(pura en griego). Se corresponde con el haplogrupo K. El 10% de los europeos pertenece a este linaje. Vivió hace 15.000 años en las planicies boscosas del Noreste de Italia, hoy en día inundada por el Adriático, y entre las estribaciones de los Alpes. Sus descendientes residen mayoritariamente en ese mismo sitio, pero también se han extendido bastante hacia el centro y norte de Europa.
Jasmine(flor en persa). Se corresponde con el haplogrupo J. Es el segundo linaje más extendido en entre la población europea, después de la prolífica Helena. También es el único linaje que tiene su origen fuera de Europa. El 12% de europeos pertenece a este linaje. Tiene su origen en el Oriente Medio hace 8.500 años. Los pertenecientes a este linaje son los descendientes de los primeros agricultores que expandieron la agricultura desde Oriente Medio.
Bryan Sykes concluye afirmando que el 80% de los Europeos descienden de sus antepasados paleolíticos.
Después se ha ido añadiendo más “hijas europeas de Eva”, menos importantes por su poca propagación por Europa. Las nuevas son:
Ulrike(Señora de todo en alemán). Se corresponde con el haplogrupo U4. Con menos de un 2% de descendientes. Originaria del frío refugio que fue Ucrania, hace 18.000 años. Sus descendientes se encuentran mayoritariamente en el este y norte de Europa, con altas concentraciones en Escandinavia y los países bálticos.
Iris(del griego, con nuestro mismo significado de arco iris). Se corresponde con el haplogrupo I. Posee menos del 2% de descendientes. Se originó aproximadamente hace 43.000 años en algún lugar de Irán, siendo los Kurdos los máximos representantes. Se propagó por Europa durante la venida de emigrantes portadores de la cultura gravetiense, hace unos 25.000 años.
JT. Hoy en día se cree que T (Tara)y J(Jasmine) vinieron “juntas” de Oriente Medio y son los principales protagonistas de la expansión de la agricultura en Europa. También va cogiendo más fuerza que estos grupos trajeron las lenguas indoeuropeas a la Europa sur y central, primero, y más tarde a la occidental y norte.
Bryan Sykes se fijó especialmente en el pueblo vasco, tradicionalmente considerado como el más antiguo de Europa. Su análisis genético no demostró nada peculiar: ofrecían los mismos resultados que los de todos los europeos, pero con una excepción notable: había representantes de los seis grupos más antiguos, pero no encontraron ninguno del más moderno, el correspondiente a Jasmine. Además son el grupo que más individuos pertenecen al linaje de Helena.
EL CROMOSOMA-Y.
ver expansión 1
ver expansión 2
El cromosoma-Y tuvo un protagonismo más reciente. Durante mucho tiempo fue considerado como un cromosoma no muy polimorfico, consistente en grandes cantidades de lo que se conoce como ADN basura, conteniendo muy pocos genes. Sólo durante esta última década muchos nuevos estudios del cromosoma-y ha mejorado sustancialmente el conocimiento sobre diferentes aspectos en lo que se refiere a su estructura y función. Su específica característica importante para el estudio de poblaciones, por la presencia de grandes áreas que no se recombinan durante la fecundación, manteniendo una transmisión únicamente patrilineal, sirve como instrumento para muchos campos de la biomedicina. El cromosoma-y también ha acumulado progresivamente cambios durante milenios debido a errores de copia y a daños por radiación.
El árbol del cromosoma-y está igualmente enraizado en un único individuo, el Adam del cromosoma-y.
ver arbol
La razón por la que el cromosoma- y es una herramienta apropiada para investigar la evolución humana reciente, genética médica y reconstruciones genealógicas, es por la singularidad que presenta entre los otros cromosomas humanos. Tiene un papel determinante en la herencia sexual, pasando únicamente de padre a hijo varón.
Los primeros estudios en este campo se iniciaron en 1985 cuando fue desvelado el primer polimorfismo en el cromosoma-y (Casanova et al. 1985).
En los 90 fueron descubiertos nuevos polimorfismos (“Fathers and sons: the Y chromosome and human evolution” Jobling, M.A. and Tyler-Smith, C. (1995)). Pero Sólo durante esta última década, arrancando con la publicación en el 1997 de Peter A. Underhill y en particular durante los últimos 5 años, ha sido cuando este campo ha tenido gran desarrollo.
Después de su comprobada gran utilidad para el estudio de poblaciones, el estudio del cromosoma-y ha ido incrementando y generalizando por grupos de estudio. Así, muchos grupos empezaron a estudiar poblaciones mediante esta herramienta y se han llegado a utilizar al menos siete diferentes nomenclaturas. Todas ellas tienen sus símbolos propios y son equivalentes. Al ser complicado seguir tan variada nomenclatura el consorcio del cromosoma-y (YCC) ha desarrollado una nomenclatura única para posteriores publicaciones.
Las dos ramas más viejas son A y B. Ambas muestran una gran distribución en el África subsahariana, aunque presentan moderadas o bajas frecuencias. La razón es la expansión que supuso sobre todo el continente africano del haplogrupo E. El 80% de africanos es descendiente directo de este linaje.
A y B están restringidos a África.
La mutación M168 representa la firma de la moderna emigración desde África al resto de continentes y África misma corroborando la teoría del origen africano reciente, excepto para los haplogrupos Ay B, restringidos en África, ya en bajas proporciones en ese continente. M168 representa, pues, el verdadero árbol coalescente.
La mayoría de ramas del árbol que representan los haplogrupos del cromosoma-y son los Hg C, D, E, y F.
La mutación que generó C parece haberse producido fuera de África, pues sus descendientes sólo habitan fuera de esta. Es probable que se generara en Asia, expandiéndose a América desde el centro y norte y a Oceanía desde el sureste.
Los haplogrupos E y D comparten su origen en África. Algunos descendientes de estos linajes permanecieron en el continente primigenio, dando lugar al haplogrupo E, el mas frecuente en africa como ya se ha dicho antes.
El Haplogrupo D se extiende por Asia aunque con bajas frecuencias, con la excepción de zonas periféricas como el Tibet, Japon e Islas Andaman.
El superhaplogrupo F, caracterizado por la mutación M89, es el padre del resto de los siguientes haplogrupos:
J y G en Oriente Medio, I en Europa, H en el sudeste de Asia. La mutación M9 da como resultado otro nuevo gran linaje llamado K. Las ramas de este gran subgrupo migraron en varias direcciones, (norte y este, principalmente) dando lugar a nuevos subhaplogrupos a partir de este haplogrupo K.
Hg J probablemente tiene su origen en Oriente Medio y es ahí donde alcanza su máxima frecuencia y diversidad, disminuyendo hacia el mediterráneo europeo, norte de África, Irán, Asia central y la India. Se divide en dos subhaplogrupos: J1 y J2, los cuales son los más comunes de nuevo en Oriente Medio. La distribución de los cromosomas-y sugiere que el hg J2 se originó en la parte norte, asociado a la expansión desde Anatolia a la parte sudeste de Europa, reflejando una expansión de agricultores desde Anatolia; y el hg J1 en la parte sur, asociado con la difusión semita(árabes, fenicios, sirios...a excepción de judios y palestinos, más parecidos a la parte anatolia, con más porcentaje de J2.)
Así del K surgen: L con grandes frecuencias en el sudeste de Asia. M restringido en Australia y Nueva Guinea. O Predominando en zonas del sur y sudeste de Asia, alcanzando el norte de China, Manchuria y algunas poblaciones siberianas. P que genera Q y R en Eurasia; Q, es característico en la población siberiana y amerindia; R es característico de la población europea y el oeste de Asia.
Por supuesto, después de esta primera expansión, cada región continental ha desarrollado su propia rama especifica; por ejemplo del linaje R resultaría R1, y R2; de R1 resulta en R1a y R1b, etc.
Además el oeste de Asia y Europa han recibido una ola adicional de genes procedentes de Africa, presumiblemente vía corredor levantino (Oriente Medio), trayendo linajes E, ausentes en la India.
VARIACIÓN EN EUROPA DE HAPLOGRUPOS DE CROMOSOMA-Y.
ver mapa
La población europea ha sido una de las que más estudios a disfrutado en los últimos años. En el año 2.000, Ornella Semino y colaboradores publicó el mayor estudio que se había efectuado hasta el momento en la población europea. Aunque Rosser y colaboradores hicieron un estudio paralelo el mismo año consiguiendo resultados similares, el estudio de Semino tuvo más eco en los medios. En el estudio de Semino resultó que más del 95% de los cromosomas-y estudiados en europeos pueden ser agrupados en 10 haplogrupos. La distribución geográfica y estimaciones de tiempo desvelaron que la mayoría de europeos desciende de dos oleadas paleolíticas y una neolítica.
La mayoría de europeos pertenecen a los Hgs R1a, R1b, I y N3, (grupos paleolíticos los cuales cubren el 73% del total) y a los Hgs J2, E3b y G (grupo neolítico que cubren el 27% restante).
La distribución de los linajes a partir de la mutación M173, sugiere que es un antiguo marcador eurasiático traído por el primer grupo de homo sapiens sapiens que entró en Europa y difundió la cultura auriñaciense hace entre 40.000 y 35.000 años. La estimación de estos linajes ha sido corroborada en estudios posteriores, tanto genéticos como arqueológicos. El 50% de europeos comparten el marcador M173 que define hg R1 y que dio lugar a los subhaplogrupos R1a, y R1b(o R1*).El hg R1b presenta altas frecuencias en el oeste europeo, siendo la Península ibérica, Irlanda, y Reino unido los máximos representantes, decreciendo hacia el este, mientras que el hg R1a presenta una frecuencia opuesta al anterior con una frecuencia máxima en el este europeo, especialmente en la población eslava.
Se atribuyó la diseminación del grupo R1a a la recolonización postglaciar, iniciada hace unos 11.000 años, desde el refugio glaciar ucraniano, aunque recientemente se ha querido atribuir la expansión por la emigración Kurgan desde el mar Caspio, siendo esta expansión entonces más reciente, según Rosser y colaboradores.
El grupo R1b cubre un área mayor que Europa, pero, como ya he dicho, el origen de este está en el occidente Europeo, revelando una diseminación postglaciar desde Iberia, lugar que sirvió de refugio para este linaje.
La mutación M170, que da lugar a hg I, revela otra expansión paleolítica, estimada de hace unos 22.000 años. Son los descendientes de hombres que llegaron desde Oriente Medio hace unos 25.000 años, asociados con la llegada de la cultura gravetiense. Los Balcanes fueron el refugio para la mayoría de este grupo.
N3 Está presente en el norte y este europeos, pero ausentes en el oeste y sur. Es también frecuente en el norte de Asia.
Los marcadores M35, M172, y M201, dan como resultado los haplogrupos E3b, J2, y G respectivamente y su frecuencia decrece desde Oriente Medio a Europa, por lo tanto estos han sido considerados los representantes de la contribución masculina de la difusión démica de agricultores venidos de Oriente Medio. Además ese flujo genético es más pronunciado en las costas mediterráneas que en la Europa continental.
En 1984 Ammerman AJ. Y Cavalli-Sforza LL. Publicaron “The Neolithic Transition and the Genetics
of Populations in Europe”, la culminación de un trabajo de más de diez años. En ella exponía el modelo de difusión neolítica. Para ello se basó en el estudio genético basado en Frecuencias de alelos en el que detectó un cambio gradual de las frecuencias de genes de este a oeste. A partir de ahí dedujo que se había producido un flujo de genes desde el Próximo Oriente en todas direcciones en época neolítica, el cual resultó coincidir con los datos arqueológicos del origen de la agricultura, llevando a pensar a Ammerman y Cavalli-Sforza que la estructura fundamental de la población europea fue determinada por la dispersión neolítica, e idearon el modelo que llamaron de difusión démica, el cual se define como el proceso específico de expansión geográfica de un grupo desde un lugar origen desencadenado por una gran innovación o innovaciones(agricultura, organización, avances de tipo militar...) que permiten o estimulan el crecimiento de una población y la expansión hacia nuevas áreas. Esta difusión se realizaría siguiendo el modelo denominado OLA EN AVANCE, el cual establece que la agricultura fue transmitida por movimientos de campesinos, que al aumentar en población a un ritmo 50 veces superior al de los cazadores-recolectores, irían extendiéndose buscando nuevas tierras para sus cultivos.

En realidad faltaba por saber la magnitud de esa expansión de genes, imposible de determinar mediante el estudio de frecuencias de alelos. Los avances en el conocimiento del genoma y en el uso tanto del ADN mitocondrial como del cromosoma-y(en los que sigue trabajando Cavalli-Sforza), han permitido conocer esta cuantía.
Así parece quedar claro que la difusión démica fue más importante en el sur, centro y este europeos, que en el oeste y norte, aunque no de manera absoluta tampoco en las del sur, centro y este, teniendo que sumar a la difusión démica la difusión cultural, esta última más importante en zonas del norte y oeste.
Como conclusión, el modelo de difusión démica explica bastante bien, a excepción de Europa, la expansión de muchas poblaciones, siendo para Europa la combinación de difusión démica y cultural, la cual puede incluir transmisión de lenguas en el plano cultural(Cavalli-Sforza, Renfrew).
En una publicación de 1.995, basándose en una selección del genoma sometida a selección a partir de HLA, Arnaiz-Villena y colaboradores postularon un origen común de peninsulares y magrebíes.
Sin embargo estudios posteriores utilizando el cromosoma-y parecían contradecir esto (Elena Bosch y colaboradores en el 2.000, Comas y colaboradores el mismo año en otro estudio). Otros estudios han ido corroborando las marcadas diferencias entre estas poblaciones (Bosch y colaboradores en el 2.001), recientemente de nuevo corroborados por un nuevo estudio del hg E3b (“Phylogeographic Analysis of Haplogroup E3b (E-M215) Y Chromosomes Reveals Multiple Migratory Events Within and Out Of Africa”, 2.004). En todos estos estudios se confirma la diferencia entre las dos poblaciones, cada una descendiente de linajes masculinos diferentes. Esta diferenciación se inició durante la primera colonización de homo sapiens sapiens en tiempos del paleolítico superior tanto en uno como en otro lado y de la cual descienden la mayoría de los pobladores actuales respectivos: por un lado los ibéricos, afines al resto de los Europeos occidentales(hg R1b), venidos a través de Asia, y por otro los magrebíes, descendientes de su propio linaje africano caucasoide (hg E3b), venidos de algún lugar del noreste de África. Así se deduce que el estrecho de Gibraltar ha servido de barrera genética entre las dos poblaciones, al menos durante un tiempo en la que ambas crecieron lo suficiente por separado.
En cuanto a la razón de los diferentes resultados de Arnaiz-Villena, esta es debida a que al seleccionar una sola parte del genoma, la naturaleza aleatoria de la deriva genética y la acción de la selección pueden producir este tipo de desviaciones; para evitarlas, la interpretación debe apoyarse en la información conjunta del máximo número posible de genes y no en una sola región del genoma.
Sin embargo, volviendo a los estudios de Bosch, Comes..., a pesar que el estrecho de Gibraltar a servido de barrera para el intercambio de genes, tanto Iberia como el norte de África han recibido flujos de pequeña magnitud hacia uno y otro lado.
Curiosamente cuando se comparan los datos del ADN mitocondrial con los del cromosoma-y, resulta que los linajes subsaharianos heredados por vía materna se hallan a una frecuencia más elevada en magrebíes e ibéricos que los linajes paternos, lo que indicaría una diferencia entre sexos en la movilidad de los individuos desde el sur del desierto del Sahara. Esta observación genética debe contrastarse con datos sociales de movilidad y comercio de esclavos. Además de eso, las poblaciones femeninas en general presentan mayor movilidad que las masculinas; Cavalli-Sforza interpreta esta movilidad como que las mujeres han tendido históricamente (y prehistóricamente)a desplazarse al lugar de origen de su compañero.
COMPOSICIÓN DESDE LA PERSPECTIVA DEL ADN MITOCONDRIAL.
Hay que decir que los porcentajes dados a continuación son orientativos, y difieren ligeramente de una publicación a otra, pero en todas ofrecen porcentajes similares. Unos son según los datos publicados en el 2.003 sobre las poblaciones del oeste del mediterráneo(University College London), otros sobre un estudio sobre pasiegos que incluían otras poblaciones ibéricas, también en el 2.003.
H y V tienen un origen en el norte peninsular y representan los haplogrupos más extendidos en Iberia, siendo H el haplogrupo más extendido en el oeste europeo, con un 49.2% de media en Iberia. Hg V alcanza un 10% en vascos, 21.6% en cantabros, y 5.2% en gallegos, pero es muy baja en el centro peninsular y Valencia, lo cual indica que la mutación de V es reciente, poco antes de la recolonización postglaciar, hace unos 17.000 años; en el norte de África está presente en saharauis(17.9%) y bereberes del sur (10%); la media en norte africanos es del 6,8%. H también alcanza un porcentaje importante en todas las poblaciones del norte de África, con un 25.6% en argelinos, 26,5% en tunicios y un 36.8% en bereberes marroquíes; esta alta frecuencia entre norteafricanos se explica mediante una expansión neolítica desde Iberia (que coincide con la de la cultura del vaso campaniforme, originada en Iberia durante el eneolítico).
J y T presentan sus frecuencias más altas en Italia con valores del 15%. Iberia muestra una distribución más heterogénea con valores del 6,6% en valencianos y de 18,7% en el sur de Portugal. El noroeste de África muestra una distribución similar de J y T que los europeos, siendo más altos en saharauis y mauritanos, estando ausente más al sur. J se subdivide en dos: J1 y J2; J1 se cree que vino a través de una ruta continental, mientras que J2 lo hizo a través de la costa mediterránea.
Hg U6(se corresponde con el E3b2*), se originó probablemente en Oriente Medio y extendió al noroeste de Africa durante el paleolítico superior hace unos 30.000 años(con altas frecuencias en mozabitas, comunidad de origen bereber que vive en Argelia(28,2%) y mauritanos(20%), disminuyendo en tunicios(4,2%) y arabes marroquíes(8%), estando ausente en argelinos). Tiene una distribución poco densa en Iberia. Está presente mayormente en la zona sudoeste peninsular, pero a bajas frecuencias, con menos del 7%, alcanzando una media del 1.8%. Está ausente en vascos, catalanes, valencianos, centro y sur de Portugal. En las Islas Canarias alcanza el 14%, debido a la clara herencia guanche. En Italia U6 sólo está presente en Sicilia( 1%). U6 se subdivide en U6a y U6b, ambos presentes en Iberia y Canarias, pero con ausencia de U6b en el norte de África, lo cual indica un movimiento de pequeños grupos hacia la península en épocas prehistóricas; así, basado en datos arqueológicos y antropológicos se cree que estos pequeños grupos primitivos fueron empujados por los portadores de la cultura capsiense, pasando los primeros (huyendo de los segundos), a Canarias y la Península Ibérica; concretamente el U6b1 tiene una frecuencia del 13% en canarios, alcanzando 0.2% en la península, lo cual indica una reciente migración hacia la península efectuada por guanches.
El hg L(que incluye el L1, L2, L3) también está presente en Iberia, pero sólo alcanza un máximo del 6.8% en el centro de Portugal, siendo en el resto de la península muy escaso. La presencia de este linaje se explica por el comercio de esclavos durante la época colonialista europea.
El resto de haplogrupos presentes, según los datos del estudio sobre las poblaciones del oeste del meditarráneo del 2.003, donde da datos de las poblaciones de Valencia, Cataluña, País Vasco, centro peninsular, Portugal, Andalucía y Galicia, van como siguen. En conjunto U, sin contar la variante U6, el centro peninsular sería el de más porcentaje con un 20%, disminuyendo hacia la periferia, con un mínimo del 9% en catalanes. K alcanza un máximo del 10% en valencianos siendo en conjunto más abundante en la periferia que en el centro peninsular, donde alcanza el mínimo de 2%; en Galicia también es bajo (3,9%). I sólo está representado en Andalucía, con un 1,9% y Portugal, con frecuencias similares. W presenta una irregular dispersión; alcanza el 5% en Cataluña, está ausente en Valencia y País Vasco y entre 1 y 2% en el resto. Por último X, con bajas frecuencias en todas las poblaciones, con un pico en el centro de Portugal del 3,6%.
COMPOSICIÓN DESDE LA PERSPECTIVA DEL CROMOSOMA-Y.
En julio del 2.004, se publicó un documento que hacía un estudio desde la perspectiva del cromosoma-y sobre la Península Ibérica. En él se comparaban 11 poblaciones ibéricas(Sevilla, Huelva, Cadiz, Córdoba, Malaga, N. Portugal, León, Galicia, Cantabria, Valencia y Castilla) con las vascas y catalanas para tener una visión más clara de la aportación genética vía paterna en la península y de paso averiguar qué procesos demográficos habían incidido en su estructura genética. Los resultados revelaron una reducida variación interpoblacional del cromosoma-y (1,2%), que apunta a una limitada heterogeneidad en la región. También se observó que ni una vieja o reciente expansión desde Oriente Medio ni del norte de África a influido en la diversidad actual del cromosoma-y en Iberia(también corroborado por restos arqueológicos), por lo que los patrones geográficos pueden ser identificados.
La población que más atención a recibido en estudios genéticos es la vasca. La creencia de que su cultura y lengua tiene un origen ancestral, ha hecho que gocen de la máxima atención de los genetistas. Y no es para menos; su composición genética presenta la proporción más alta del haplogrupo R1b con un 89%(que coincide con la expansión del hg H del ADN mitocondrial, igualmente alto en vascos) y a penas posee en su composición genética haplogrupos de origen neolítico-anatolio(J, G, E3b). La composición genética también demuestra un cierto aislamiento y endogamia. Sin embargo no es una población aislada y hoy en día queda demostrado su relación con sus poblaciones vecinas ibéricas.
Los pasiegos es un grupo peculiar dentro de las poblaciones ibéricas, ya que recientemente se ha revelado un alto porcentaje del hg E3b de origen norteafricano con un 41% de individuos pertenecientes a este linaje (“Phylogeographic Analysis of Haplogroup E3b (E-M215) Y Chromosomes Reveals Multiple Migratory Events Within and Out Of Africa”, 2.004), demostrando ser una población muy heterogénea compuesta por grupos de diferente origen; uno peninsular y otro bereber seguramente venidos durante la invasión musulmana.
El resto de poblaciones, a excepción de la catalana, muestra una composición más heterogénea, fruto de las sucesivas invasiones recibidas a lo largo de la historia de la Península, aunque las aportaciones de estos linajes son mucho más modestas, pues la población ibérica ya era abundante desde el neolítico y estos eran a su vez los descendientes de los primeros pobladores portadores de la cultura auriñaciense.
Según este estudio el 61,4% pertenecen al linaje más antiguo, el denominado R1b, el más común en el oeste europeo y que define en más del 50% de los cromosomas-y ibéricos, siendo el más frecuente en todas las poblaciones.
Los linajes de origen africano alcanzan un 10,8% del total, alcanzando en algunas poblaciones altas frecuencias como es el caso de Galicia con un 46% y Malaga con un 30.6%. Curiosamente en el resto de poblaciones andaluzas el porcentaje no es muy alto, entre un 3,6% en Cadiz y un 13,5% en Huelva. N.Portugal 15.5%, León 10%, Valencia 16,2% Cantabria 12,9%, Castilla 4,8%, en catalanes no se ha registrado ningún linaje norteafricano y en vascos un 2,2%. Están relacionados por la llegada de grupos norteafricanos durante la invasión musulmana.
Por otra parte los haplogrupos I alcanzan altas proporciones en Andalucia y levante, aunque es bastante alto en Castilla (33,3%)y comparable a los porcentajes de Cerdeña, donde el porcentaje es el más alto de toda Europa. El subhaplogrupo I1b2 está relacionado con un grupo que presumiblemente usó Iberia como refugio durante la última glaciación, aunque su aporte a estas poblaciones es pequeño. También tiene un porcentaje en vascos del 8,8%.
El haplogrupo J tiene una media del 6,2%. Muestra una tendencia decreciente desde el sudoeste hacia Cataluña, con 17,9% en Andalucía, alrededor del 5% en León, Galicia, y Cantabria, 9,7% en Valencia siendo casi inexistente en Cataluña y País vasco. Está relacionado con la expansión neolítica originaria de Oriente Medio.
El grupo G no es considerado importante, por su baja frecuencia en las poblaciones, aunque está relacionado también con la expansión neolítica. Tiene un porcentaje entre el 6 y el 8% en N. Portugal, León, Cantabria y Cataluña, mientras que en el sur está en muy bajas proporciones y es inexistente en vascos, gallegos, castellanos y valencianos.
El estudio concluye resaltando la poca distancia genética entre las poblaciones ibéricas, incluida la vasca, una vez se quitan los datos de aportes genéticos minoritarios, que son las que crean cierta distancia genética.
A pesar de la compleja historia que ha sufrido la Península Ibérica el estudio demuestra la importante aportación de las poblaciones paleolíticas, la reducida estructura genética entre las poblaciones ibéricas y, por tanto, la gran afinidad de todas ellas entre sí, genéticamente hablando.
FENOTIPOS.
El primer genetista del mundo fue Mendel. Sus descubrimientos le llevaron a postular las tres leyes que llevan su nombre pudiendo así conocer cómo se producía la herencia genética. Así, él demostró que los caracteres se transmitían independientemente a través de unidades hereditarias que eran por naturaleza discontinuas; además estos caracteres no se fundían para siempre en la siguiente generación sino que se segregaban en la nueva formación de los gametos(espermatozoide y óvulo), es decir, un modelo de herencia no mezclada. Cuando el individuo produce sus células sexuales en estos sólo viajan la mitad de sus genes, mezclandose con la otra mitad del otro progenitor durante la fecundación. El nuevo individuo resulta así con una dotación genética doble cuyos caracteres hereditarios se regulaban mediante alelos dominantes y recesivos.
Luego vino la teoría de la mutación de Hugo de Vries, cambio espontáneo y al azar el cual era un cambio genético de envergadura, creando nuevas especies si las mutaciones eran las apropiadas para sobrevivir en el medio.
Si se suma la adaptación al medio mediante la selección natural, ya tenemos una idea de cómo se producen cambios en la morfología de los individuos y la diferenciación de unos grupos de otros.
Entonces además de depender de nuestra herencia genética, dependemos de mutaciones que se añaden a nuestro acerbo genético y que deben ser aptas para permitirnos la vida en un determinado medio. Además nuestra morfología dependerá de dietas, enfermedades, que afectarán a nuestra salud y desarrollo individual.
Se podría pensar que los diferentes haplogrupos coinciden con las razas actuales. Pero el problema es que las mutaciones son aleatorias tanto para la mutación en si como para el tiempo en el que se produce, por lo que dos poblaciones con fenotipos diferentes pueden pertenecer al mismo haplogrupo y parecer de mundos diferentes. En esto hay muchos factores que intervienen, como ya se ha dicho arriba.
Un ejemplo claro de esto es el nuestro: Los europeos somos descendientes de nuestros antepasados paleolíticos, fundamentalmente de los portadores de la cultura auriñaciense a Europa, cuyo representante en el registro fósil es el hombre de cromañon. Éste era de elevada estatura, entre 1,70 y 1,85 m, caderas estrechas, dolicocéfalos y de cara ancha.
Pero durante la última glaciación vemos una evolución de éste tipo, viendo como tiende a ser más bajo. Así, observando el registro fósil, tenemos al hombre de Combe-Capelle, correspondiente a la cultura perigoriense, de 1,63m y dolicocéfalo, ya empieza a mostrar rasgos de la raza mediterránea que tendrá su difusión en tiempos del mesolítico y neolítico por toda Europa. Lo mismo se puede afirmar sobre Los hombres de Chacelade, que se consideran descendientes del de Combe-Capelle.
La razón de esto se cree que se debe a diferentes factores:
Una adaptación a un ambiente extremadamente frío, en donde los individuos “redondos” o menos alargados, mantienen el calor mejor que los individuos “alargados” o altos. Esto no implica una menor robustez, siendo las poblaciones más bajas pero bien construidas.
Para el antropólogo Lionel Sims la pérdida de robustez se debe a una etapa en la que desaparecieron los grandes animales, cuya caza implicaba un trabajo en equipo, camuflaje y experiencia, con una vida nómada, sin enfermedades típicas de las poblaciones sedentarias, hacía de esta una vida más excitante y sana; al depender más de la caza de los pequeños animales, más difíciles de cazar, y al verse envueltos en un rápido cambio en su ambiente, el paso fue más bien traumático; al principio el paso a una vida sedentaria al adoptar la agricultura resultaría aburrida para individuos acostumbrados a una actividad más excitante; eso sumado al desarrollo de enfermedades típicas del sedentarismo y desconocidas para nuestros antepasados hasta entonces, contribuyó a un empeoramiento de la salud y, por tanto, dando lugar a individuos con esqueletos más gráciles, afectando también a la altura.
El color muy claro de los pobladores de las regiones del norte de Europa se debe, según Marvin Harris, a que pueblos de fenotipo mediterráneo producían alimentos agrícolas, derivados lácteos, y obtenían la vitamina D del sol. Pero al comenzar su lenta ascensión hacia el norte, sufrieron un déficit de vitamina D (que se obtiene normalmente del pescado marino) y de calcio que hizo que la selección natural favoreciese la piel blanca, por su mayor receptividad frente a las radiaciones solares, y también por su mayor capacidad para producir la enzima llamada lactasa que posibilita la ingestión de grandes cantidades de leche fresca, alimento básico en los territorios nórdicos. Así, la supervivencia de hijos de piel clara, más fuertes y más sanos por lo general que los hijos morenos en ambientes de poca radiación solar con déficits alimenticios, unido a la alta mortalidad infantil de esas épocas, dio como resultado a una mayor proporción de rubios en las poblaciones nórdicas. Para Harris la mayor proporción del tipo nórdico se produjo hace 6.000 años, pero en realidad se puede asegurar que entre las poblaciones paleolíticas (caucasoides o no) ya habían individuos rubios, aunque no fueran ni mucho menos mayoritarios con respecto a los morenos.
Para Henri V. Vallois, los nórdicos son "un conjunto heterogéneo que corresponde a la despigmentación independiente de grupos diferentes". Este punto de vista concuerda con la diversa composición genética que presentan las poblaciones del norte europeo, y que sin embargo son la máxima representación del tipo nórdico.
Viendo la variedad de fenotipos tan grande dentro de un mismo haplogrupo nos damos cuenta que los cambios morfológicos suelen producirse más rápido que las mutaciones que dan lugar a nuevos haplogrupos, por lo que no se puede relacionar haplogrupo con fenotipo.
LENGUAS.
Cavalli-Sforza cree que los linajes del cromosoma-y pueden estar asociados con los mayores grupos lingüísticos del planeta. Y la verdad es que viendo la distribución de las principales familias lingüísticas del mundo y comparandolas con los haplogrupos correspondientes, vemos que lo que era imposible con los fenotipos, no lo es tanto con las lenguas. Aquí si que vemos cierta correlación para las lenguas afroasiáticas, urálicas, amerindias(antes de la llegada de europeos), etc, aunque con excepciones como la del grupo indoeuropeo, en la que hay controversia sobre sus orígenes.
Los hablantes de las lenguas indoeuropeas abarcan grupos humanos de origen lo suficientemente separado en el tiempo para que todos hablaran unas lenguas tan parecidas entre sí que se pudieran incluir en la misma familia. Así tenemos a nuestros ancestros que habitaban el occidente europeo antes de la última glaciación, y los que habitaban el este. Ambos recolonizaron Europa después de la retirada de los hielos y ambos utilizaban su propio lenguaje. Una separación de más de 20.000 años no puede dar lugar a grupos que hablaran lenguajes parecidos que se conservaran igual durante tal espacio de tiempo. Por lo que la única alternativa que nos queda es la de una sustitución de unas lenguas por otras.
Tanto el genetista Cavalli-Sforza como el arqueólogo Colin Renfrew(siguiendo el modelo de ola en avance, basandose en el registro arqueológico y ahora también en el genético), creen que las lenguas indoeuropeas fueron traídas por los primeros grupos neolíticos, portadores de la agricultura y la ganadería desde su ubicación original, en Anatolia.
El filólogo finlandés Kalevi Wiik se ha basado en estos últimos descubrimientos genéticos para lanzar su teoría según la cual entre hace 23.000 y 8.000 años Europa estaba dividida en tres regiones; dos llamada Ba(protovascas) al oeste, y U (uralicas) al noreste, poblada por cazadores de grandes animales que abundaban en esas épocas. Una tercera zona denominada X que corresponde a la zona centro y sur de Europa y que estaba poblada, según él, por cazadores de pequeños animales, zona que estaba fragmentada en varias lenguas que no han sobrevivido.

Entonces hace unos 5.500 años, hubo un cambio radical que hizo que las zonas Ba y U entraran en recesión; este fue la desaparición de los grandes animales en esas zonas, mientras que en la zona X empezaron a adoptar las formas de viva Neolítica, domesticando animales y desarrollando la agricultura, que les hizo más fuertes económicamente a la vez que desarrollaron sistemas de organización complejas, creciendo en número. Es aquí donde Kalevi Wiik defiende que desde los Balcanes y Grecia ( los exportadores del Neolítico, según él) se expandió el indoeuropeo, sirviendo como lengua vehicular para los habitantes de la zona X, reemplazando las lenguas de la zona X y convirtiendo más tarde a los menos exitosos habitantes de la zona Ba y U.

Finalmente, justo antes de la conquista romana, en las zonas más occidentales, todavía habrían muchos hablantes de lenguas Ba que habrían adoptado la agricultura y ganadería sin perder sus lenguas, aunque con gran influencia indoeuropea, como la de los ligures, las lenguas ibéricas y/o euskéricas, de los pictos o cruithin en Escocia e Irlanda, y de los pridyn en Gales,

hasta que finalmente desaparecieron por conquista de romanos o gaélicos, con la excepción del Vasco.

Para algunos lingüistas, entre los que se encuentran Theo Vennemann, la lengua vasca es la última representante de una familia de lenguas extendidas por todo el occidente europeo en épocas antiguas y que fueron sustituidas por las lenguas indoeuropeas, portadas por grupos originariamente venidos de Anatolia y sudeste europeo.
Bibliografía.
Laboratorio de Evolución, Facultad de Ciencias, Universidad de la República(Uruguay):
http://evolucion.fcien.edu.uy/Diapositivas/GenesHumanos4d.pdf
De la web de Cristina Díaz. Apartado “de dónde venimos”(La superexplosión del Toba): http://www.mundofree.com/origenes/sapiens/donde.htm
Glosario de Términos Genéticos: http://www.genome.gov/sglossary.cfm
Red Cívica Americana. El Árbol genealógico de la Familias humana:
http://www.redcivica.com.uy/archivos/html/revista/tomoIII/El%20arbol%20genealogico.htm
Tesis de Stèphanie Plaza. Anàlisi de la diversitat del genoma mitocondrial en poblacions humanes:
http://www.tdx.cesca.es/TESIS_UPF/AVAILABLE/TDX-0709104-123956//tspp1de1.pdf
Técnicas de análisis del ADN en genética forense: ANÁLISIS DEL ADN EN GENÉTICA FORENSE
http://members.fortunecity.es/robertexto/archivo7/adn_forense.htm
Patología y mecanismos de los trastornos genéticos:
http://ventanadelavida.jesussaveus.com/apuntesmedicina/genetica.htm
Orígenes de los Humanos Modernos: ¿Multiregional o Fuera de África?
Por Donald Johanson:
http://www.actionbioscience.org/esp/evolution/johanson.html
Juan Luis Arsuaga-EVOLUCIÓN HUMANA-C.N.I.C.E.:
http://www.cnice.mecd.es/tematicas/evolucion/2002_07/2002_07_01.html
Tesis de Siiri Rootsi: HUMAN Y-CHROMOSOMAL VARIATION IN EUROPEAN POPULATION:
http://www.tymri.ut.ee/PhD/2004/SRootsi_thesis.pdf
British teacher finds long-lost relative: 9,000-year-old man
http://www.standardtimes.com/daily/03-97/03-09-97/a09wn056.htm
Oetzi, el hombre de hielo http://news.bbc.co.uk/hi/spanish/science/newsid_3231000/3231879.stm
Las siete hijas de Eva.
Bryan Sykes http://usbiver.tripod.com/hija.html
mtDNA haplogroup of L. David Roper
http://www.roperld.com/mtDNA.htm
The Human Family Tree: 10 Adams and 18 Eves By Nicholas Wade
http://www.ramsdale.org/dna7.htm
World haplogoups maps, J. D. Mcdonald http://www.scs.uiuc.edu/~mcdonald/WorldHaplogroupsMaps.pdf
Inferring Human History: Clues from
Y-Chromosome Haplotypes
P.A. UNDERHILL http://hpgl.stanford.edu/publications/Underhill_2004_p487-494.pdf
The Molecular Dissection of mtDNA Haplogroup H Confirms That the
Franco-Cantabrian Glacial Refuge Was a Major Source for the European
Gene Pool
Alessandro Achilli y colaboradores, http://evolutsioon.ut.ee/publications/Achilli2004.pdf
Demic Diffusion as the Basic Process of Human Expansion. http://hpgl.stanford.edu/publications/Examining_2003_chapter7.pdf
Expansión de mtDNA mundial según J. D. Mcdonald. http://www.mcdonald.cam.ac.uk/genetics/mtDNAworld/one.html
Phylogeographic Analysis of Haplogroup E3b (E-M215) Y Chromosomes Reveals Multiple Migratory Events Within and Out Of Africa, 2.004. http://www.familytreedna.com/pdf/hape3b.pdf
Reduced genetic structure of the Iberian peninsula
revealed by Y-chromosome analysis: implications for
population demography. http://hpgl.stanford.edu/publications/EJHG_2004_v12_p855.pdf
A Signal, from Human mtDNA, of Postglacial Recolonization in Europe. http://www.oxfordancestors.com/papers/mtDNA01%20Postglacial.pdf
mtDNA Analysis in Ancient Basque Populations http://www.journals.uchicago.edu/AJHG/ journal/issues/v69n4/012962/012962.web.pdf
Alu insertion polymorphisms in NW Africa and the Iberian Peninsula:
evidence for a strong genetic boundary through the Gibraltar Straits.
http://batzerlab.lsu.edu/Publications/ Comas%20et%20al.%202000%20Hum%20Genet.pdf
Mitochondrial DNA transit between West Asia and North Africa
inferred from U6 phylogeography. http://www.biomedcentral.com/content/pdf/1471-2156-4-15.pdf
The Genetic Legacy of
Paleolithic Homo sapiens
sapiens in Extant Europeans: A
Y Chromosome Perspective. http://hpgl.stanford.edu/publications/ Science_2000_v290_p1155.pdf
El enigma de la esfinge, Juan Luís Arsuaga.
Enciclopedia Universal Multimedia ©Micronet S.A. 1999/2000.
Comentarios
 Pulsa este icono si opinas que la información está fuera de lugar, no tiene rigor o es de nulo interés.
Pulsa este icono si opinas que la información está fuera de lugar, no tiene rigor o es de nulo interés.
Tu único clic no la borarrá, pero contribuirá a que la sabiduría del grupo pueda funcionar correctamente.
Si te registras como usuario, podrás añadir comentarios a este artículo.


Saludos Onnega:
El ADN nuclear sí que se ha estudiado:
"La carrera por descifrar la secuencia completa del genoma humano y lograr la localización de los genes en los cromosomas fue ganada por la técnica automatizada del emprendimiento privado, que el 6 de abril de 2000 anunció el primer borrador"
http://www.biotech.bioetica.org/ap13.htm
En el ADN nuclear de cualquier célula de un individuo se encuentra toda la información de este.
La mitad de los genes en la fecundación la aporta el espermatozoide, la otra mitad el óvulo. Es dificil trazar la historia de poblaciones a partir de este ADN nuclear que se recombina en cada fecundación.
Partes del cromosoma-y no se recombina durante la fecundación, lo mismo que el ADN mitocondrial, y sólo mutan aleatoriamente por fallos de copia muy de vez en cuando. Es por ello por lo que sirve para el estudio de poblaciones.
Quizá el neandertal era de nuestra misma especie, es decir, no era un homínido diferenciado, pero todas las pruebas apuntan a que no se mezcló con nosotros.
No creo que los científicos del CSIC y la universidad de Barcelona sean tranposos, sólo aportan pruebas científicas. Sería muy interesante que los que creen que venimos del neandertal aportaran pruebas para defender su posición. La interpretación de los fósiles puede alimentar la controversia pero la genética es una ciencia exacta (o casi). Si se decantan claramente por la teoría del origen reciente es porque las pruebas genéticas son sólidas:
"Madrid, 14 de febrero, 2005.- Un equipo de investigadores españoles, dirigidos por el paleobiólogo del Consejo Superior de Investigaciones Científicas (CSIC) Antonio Rosas, en colaboración con el investigador de la Universidad de Barcelona Carles Lalueza, ha alcanzado un logro científico sin precedentes al conseguir extraer y secuenciar por primera vez ADN mitocondrial de restos neandertales en la Península Ibérica. Los fósiles analizados en el estudio proceden de la cueva asturiana de El Sidrón y tienen 43.000 años de antigüedad."
"El análisis de la secuencia de ADN obtenido ha permitido a los investigadores establecer que las distintas poblaciones de neandertales que habitaron Europa desde la Península Ibérica hasta el Cáucaso eran genéticamente similares. Esta conclusión, en contra de lo que algunas hipótesis previas hacían suponer, permite situar un origen común de los neandertales hace 250.000 años. Esta fecha coincide con la aparición de la morfología de neandertal clásico y concuerda asimismo con un posible evento de especiación para el origen de este Homo neanderthalensis."
http://www.csic.es/prensa/noticias2005/febrero05/14febrero2005.html
Otro sitio:
http://www.diariomedico.com/edicion/noticia/0,2458,594450,00.html
Lo malo de estos estudios es que si no se es un especialista en la materia es muy dificil interpretarlos, es muy fácil crear confusión y dar a entender lo contrario de lo que dicen los datos.
En primer lugar la mutación que define E3b no llegó de áfrica. La mayoria de los que son E3b en la península proceden de la europa central.
E3b es una mutación antiquísima( 25000 años), una parte de ellos entro en los balcanes y centroeuropa hace miles de años dando lugar a los E3b europeos, otra parte llegó al norte de áfrica, los antes llamados E3b2:
http://freepages.genealogy.rootsweb.com/~dgarvey/DNA/hg/YCC_E3b.html
Sólo una pequeña parte de los E3b peninsulares son en realidad de origen bereber.
Otra cosa: el cromosoma Y al que pertenecen estas mutaciones no se recombina, en otras palabras: si un vikingo llega a irlanda en el siglo IX y deja su cromosoma Y desde entonces, sus descendientes varones tendrán un cromosoma y tipicamente vikingo aunque sólo tengan un antepassado vikingo y 9999 antepassados indígenas irlandeses. Si por azar( deriva), esos descendientes predominan en una comarca irlandesa algunos afirmarian que son descendientes de vikingos, pero al mirar sus otros cromosomas( 22 más el mitocondrial) veríamos que son tipicamente irlandeses( provienen de sus 9999 antepassados irish).
Los Pasiegos no son en un 40% bereberes como sugieren algunos, porque al mirar el resto de variaciones genéticas en el resto de cromosomas se revelan como tipicamente europeos.
Que haya un 5% de cromosomas norteafricanos en italia, portugal o francia no significa que sean descendientes en un 5% de norteafricanos.
Es muy fácil manipular estos debates y intentar dar una impresión falsa con fines políticos o ideológicos.
La peninsula ibérica es globalmente una zona tipicamente europea, la mayor parte de ella tiene afinidades con los pueblos del norte y centro de italia y sur de francia. La franja atlántica norte-noroeste tiene mayor afinidad con poblaciones atlánticas europeas( puro determinismo geográfico). No obstante la variación entre poblaciones europeas es pequeña.
dasagis08 de abr. 2008Información eliminada por el Administrador Silberius
dasagis11 de abr. 2008Información eliminada por el Administrador Silberius
Hay 4 comentarios.
1